
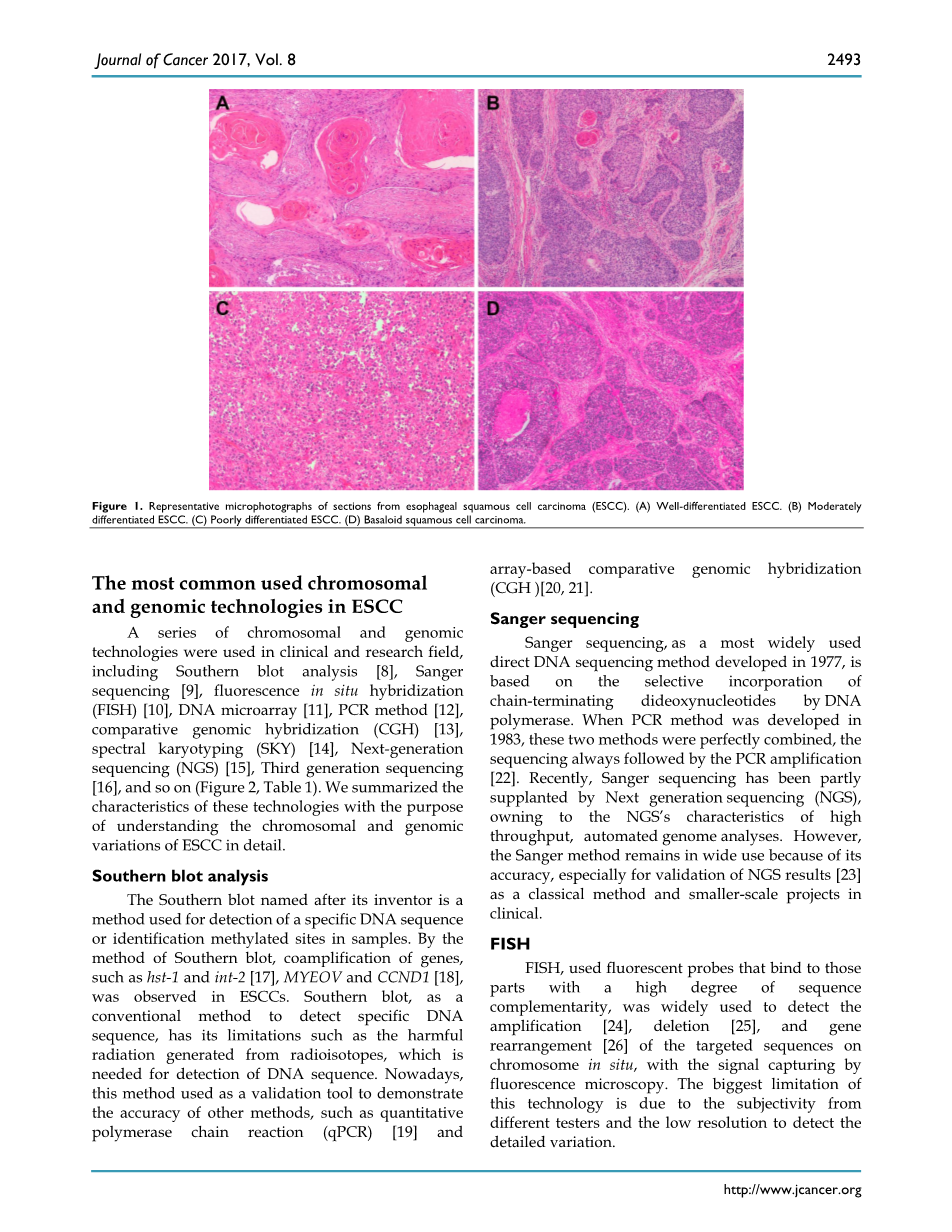
英语原文共 9 页,剩余内容已隐藏,支付完成后下载完整资料
食管鳞癌染色体和基因组变异:关于技术,应用和探索的综述
摘要
食管鳞状细胞癌(ESCC)是世界上最常见的预后不良恶性肿瘤之一。 预后不良是由于确诊时已经是晚期阶段,也缺乏能为患者确定治疗方案的显著分子生物标志物。 由于癌症是一种由于基因组不稳定性和遗传变更累积导致的疾病,染色体和基因组技术的发展迅速,用在基因水平诊断患者。 在这篇综述中,我们总结了ESCC染色体和基因组水平的研究进展以及多种技术的应用。
介绍
食管癌是世界第八最常见的癌症,第六常见的导致死亡的癌症。2012年大约有456000个新增病例,4000000人死亡。但根据中国最新的癌症统计数据,食管癌是男性第三普遍的癌症,女性排名第五。当涉及死亡率时,食管癌是致使癌症死亡的第四大主要原因。 食管癌可分为食管鳞癌(ESCC)和食管腺癌(EAC),在组织病理学,流行病学和分子层面它们是两种完全不同的亚型。全球范围内ESCC大约占食管癌的90%,而且ESCC患者的5年生存率虽然在过去十年有所改善,但依旧普遍较低。许多患者在诊断时表现为淋巴结转移和肿瘤侵袭邻近器官,缺乏有效的化疗方法治疗ESCC患者,这两者均对ESCC预后不良有影响。目前的临床分期方法在有效分层治疗方案的能力方面有限。最近提出的精密医药耦合概念建立了临床病理指标,并通过分子分析建立了预防、诊断、预测和治疗策略,这些都是针对每个病人的需求量身定制的。这个概念让我们所有人都可以使用个性化分子技术,更容易治愈疾病。在此综述中,我们总结了近几十年来在ESCC临床和研究领域中使用的一系列染色体和基因组技术,帮助我们了解ESCC的染色体和基因组特征和变异,并为我们提供与分子水平分析水平有关的精准医疗的直接认知。
食管癌中最广泛使用的染色体和基因技术
一系列的染色体和基因技术被用于临床和研究领域,包括印迹分析,桑格测序,荧光原位杂交(FISH),DNA微阵列,PCR方法,比较基因组杂交(CGH),光谱核型分析(SKY),下一代测序(NGS),第三代测序等等。我们总结了这些技术的特点,目的是详细了解ESCC的染色体和基因组变化。
Southern印记分析
以其发明者命名的Southern blot是一种用于检测样品中特定DNA序列或鉴定甲基化位点的方法。采用Southern blot方法,在ESCC中观察到hst-1和int-2、MYEOV和CCND1等基因的扩增。作为一种检测特定DNA序列的常规方法,Southern blot有其局限性,如放射性同位素产生的有害辐射,但这是检测DNA序列所必需的。现在,这种方法被用作验证工具,以证明其他方法的准确性,如定量聚合酶链反应(qPCR)和基于array的比较基因组杂交(CGH)。
桑格测序
Sanger测序法是在1977年发明的一种最广泛使用的直接DNA测序方法,它的基础是DNA聚合酶选择性终止双脱氧核苷酸链。当PCR法在1983年发明时,这两种方法完美的结合在一起,测序后总是进行PCR扩增。最近,Sanger测序已经部分地被下一代测序(NGS)取代,因为NGS具有高通量,自动化的基因组分析的特性。然而,Sanger方法由于其对NGS结果验证的准确性,仍作为一种经典的方法被小规模的临床项目广泛使用。
FISH
FISH使用荧光探针与那些具有高度序列互补性的部分结合并通过荧光显微镜捕获信号,被广泛用于检测染色体原位靶序列的扩增,缺失和基因重排。 这项技术的最大局限是不同测试人员的主观性和用低分辨率来检测详细变化。
DNA微阵列
DNA微阵列提供了一种高通量的基因组学方法,可以系统地筛选染色体变异,并可用于同时测量大量基因的变化或对基因组的多个区域进行基因型评估,以评估肿瘤的异质性。然而,DNA微阵列设计和制造的复杂性限制了这种技术的使用。
PCR方法
PCR方法是一种广泛使用的技术,用于扩增特定的DNA或cDNA序列以产生数千至数百万个拷贝,是许多应用的重要基本方法,例如PCR-测序,PCR-限制性片段长度多态性(RFLP ),PCR-单链构象多态性(SSCP),突变扩增系统(ARMS)-PCR等。 PPCR的主要局限性是在指定引物之前需要目标序列信息。此外,由于PCR是一种高灵敏度的技术,任何形式的样品污染都可能产生误导的结果。
PCR测序
这种组合技术被广泛用于科学研究和分子病理学检测在一个或多个基因的特定区域的单核苷酸多态性(SNP),突变和基因融合。
PCR-RFLP
PCR-RFLP是一种利用同源DNA序列变异的技术。 扩增PCR后,用限制性酶分解DNA产物,并通过凝胶电泳按长度分离所得的限制性片段。 RFLP分析是基因组定位,遗传疾病基因定位和疾病风险测定的重要工具。
PCR- SSCP
SSCP定义为在特定实验条件下不同的碱基序列引起的的相同长度的单链核苷酸序列的构象差异。 这种特性使得序列可以通过凝胶电泳区分,凝胶电泳可以根据不同的构象来分离片段。
ARMS-PCR
这项技术的基础是在适当条件下,有不匹配的3 #39; -残留物的寡核苷酸在PCR中不作为引物发挥作用。 ARMS-PCR简单,快速,可靠,可以直接分析任何感兴趣的位点,提供充足的序列数据。
实时qPCR
实时qPCR是基于常规PCR的技术,用于监测PCR过程中靶向DNA分子扩增的信号。用于检测实时qPCR产品的两种常用方法是:(1)插入任何双链DNA的非特异性荧光染料,如SYBR Green方法,这种方法是用于检测基因拷贝数变更(CNA)的低成本且准确的方法,包括扩增和删除的拷贝数; (2)由带有荧光报道分子标记的寡核苷酸组成的特异性DNA序列探针,只有在探针与其互补序列杂交后才能被检测到,如TaqMan探针法。一系列的TaqMan分析被设计来检测SNP ,CNA 和基因突变。这种方法含有特异性DNA探针,可以提高检测的特异性和灵敏度,适用范围更广,比SYBR Green方法准确,但成本更高。
液滴数字PCR
数字聚合酶链式反应可以同时扩增数千个样品,每个样品都位于乳液中的一个单独的液滴中。 这可用于在DNA样本中量化突变等位基因或特定基因的拷贝数。
CGH
CGH是一种强大的方法,可以在一个杂交实验中对肿瘤细胞的整个基因组进行DNA CNAs检测,并且与较传统的FISH细胞遗传学分析技术相比,分辨率有所提高,FISH技术受显微镜分辨率的限制。目前,这项技术与DNA微阵列完美结合,可以检测不平衡的染色体异常。
SKY
SKY用不同颜色将生物体中的所有染色体对同时显示出来,检测或定义基因组变化,如染色体衍生物或染色体重排。但是,它检测复杂的重排能力有限。
NGS
对低成本测序的高需求推动了NGS技术的发展,将测序过程并行化,同时生成数千或数百万个序列。随着NGS的广泛使用,各种疾病的遗传景观已有报道。然而,如果没有生物信息学,花费大量资金从NGS获的的结果可能是毫无价值的。此外,从NGS获得的序列需要用其他经典方法进行验证。
第三代测序
第三代测序的特点是不需要PCR扩增的单分子测序。这允许更长的读取,更快的测序速度和更高的准确性。由于长序列的直接测序,很容易进行数据分析而不需要结合基因序列。而且,RNA和甲基化的DNA位点可以直接测序。
ESCC中的染色体变异
染色体变异是染色体DNA的一系列缺失,多余或不规则部分。它可以来自一个或多个染色体或染色体片段的非典型数目的染色体或结构异常。
ESCC中的染色体变异
染色体变异是染色体DNA的一系列缺失,额外或不规则部分。它可以来自一个或多个染色体或染色体片段的非典型数目的染色体或结构异常。
染色体臂的增加和缺失/损失
在1p,1q,2p,3q,5p,6p,7p,7q,8q,9q,11q,12p,14q,15q,16p,16q,17q,18p,19q,20p,20q ,22q和Xq上发现最常见的染色体基因扩增,而最常见的缺失涉及3p,3q,4p,4q,5q,6q,7q,9p,13p,13q,18q和19p 。 12p的增加表明食管切除手术后预后不良。 但是,值得指出的是,由于该时期的技术有限,这个结论应该通过进一步的研究来证实。
近年来,基于阵列的高分辨CGH(aCGH)已被用于通过定义各种癌症的复发增益和损失来识别目标癌基因和肿瘤抑制基因(TSG)。 对ESCC样品的研究揭示了在3q26.32-33, 3q27.1, 7p11, 7p22.3, 8p11.23, 8q21.11, 8q24.21, 11q13.2-11q13.3, 11q22, 12p12.1, 12q15–q21.1, 13q22.1, 14q11.2, 14q13.3, 18q11.2, 和19q13.11–q13.12中的复发性,高水平扩增 ,以及1p15.4, 2q22.1-22.2, 3p14.2, 4p16.1-p15.1, 4q34.3–q35.1, 5q12.1, 6p22.1, 9p21.3, 9p24.1, 13q14.2, 14q12, 和22q13.1中纯合子的缺失。11q13.2的增加和7q34和18q21.1-q23的缺失与预后不良有。
染色体结构变异
当染色体结构发生改变时,会出现几种非典型模式,如缺失,重复,易位,倒位,插入等。 它们通常导致某些类型的恶性肿瘤的发展趋势加强。 Caixia Cheng等分析来自31个ESCC的全基因组测序(WGS)数据以预测体细胞结构变化并确定拷贝数变化。 他们发现缺失和易位是占主导地位的SV类型,16%的缺失是复杂的缺失。 Chromothripsis,kataegis和断裂融合桥(BFB)被认为是导致55%的ESCC发生染色体局部错位的原因。
ESCC中的基因变异
遗传变异是建立在基因库中等位基因变异的基础上的。基因变异会导致表型变异,如果该DNA序列的变异导致了由该DNA序列编码的蛋白质中氨基酸的顺序的差异。我们关注的是基因CNAs和ESCC中的的突变
基因突变
基因突变是导致基因功能改变或丧失的重要机制。在ESCC中,最常发生突变的基因包括TP53、PIK3CA、BRCA2、EGFR、NRF2、CDKN2A等,是用传统的方法,如pcr -测序或PCR-SSCP检测到的。ESCC的完整的遗传景观仍然不完整,很可能其他的基因也可能在这种疾病及其进展中发挥作用。测序技术的最新进展克服了过去的规模限制,在单一样本中可以识别数千个突变。中国研究人员对ESCC患者进行全基因组测序或全基因组测序,希望能够定义ESCC的突变景观,为了解食管肿瘤提供重要的分子基础。传统方法发现的突变也被NGS检测到,并且在ESCC中首次发现了一些新的突变基因。在表2中,我们在两个NGS的研究中对经常突变的基因进行了分析。最常见的突变基因为TP53、TTN、MLL2、CDKN2A、PIK3CA、NOTCH1、NFE2L2、EP300、ADAM29、FAM135B等。这些基因主要参与包含表观遗传过程的途径(MLL2, EP300, CREBBP, TET2);细胞周期(TP53, CCND1, CDKN2A, FBXW7);而NOTCH (NOTCH1, NOTCH3), WNT (FAT1, YAP1, AJUBA)和受体酪氨酸激酶磷酸肌肽3-激酶信号通路。PIK3CA, EGFR, ERBB2
Ling Zhang等讨论了ESCC的突变特征。A信号的特征是在TpCpX三核苷酸突变中Cgt;G, Cgt;T, Cgt;A,与胞嘧啶脱氨酶APOBEC家族的突变有关。B信号的特征是在XpCpG三核苷酸突变中中Cgt;T增加,因为自发性5-甲基胞嘧啶脱氨率升高。参与细胞周期和凋亡调控的基因在99%的病例中发生突变,在ESCCs中约63%的情况下观察到调控组蛋白修饰的基因突变。
为了确定一些显著变异基因的更深层次的临床意义,研究人员将注意力集中在有害周期性突变的基因的预后价值上。EP300,TET2,FAM135B突变与较低的生存率有关。然而,NOTCH1突变比那些没有有害突变的个体有更好的结果。这些结果需要在今后的研究中进一步验证。
基因CNAs
基因扩增
基因扩增是导致原癌基因激活的主要原
剩余内容已隐藏,支付完成后下载完整资料
资料编号:[464146],资料为PDF文档或Word文档,PDF文档可免费转换为Word
您可能感兴趣的文章
- 纳米材料介导镉污染土壤原位修复的机理及研究进展外文翻译资料
- 纳米零价铁磁洗受镉污染水稻土壤的有效性验证外文翻译资料
- 镉对水稻叶片生长和养分运输的短期影响外文翻译资料
- 间接竞争ELISA的研制及可视化基于金纳米棒生长的多色酶联免疫吸附试验玉米赤霉烯酮的测定外文翻译资料
- 铅(Pb)诱导的小麦根系生化和超微结构变化外文翻译资料
- 用蚕豆根尖微核试验评估污水灌溉区和生物修复区土壤的遗传毒性外文翻译资料
- 镧在红土壤中的生物累积和其对玉米幼苗生长的影响外文翻译资料
- 铈对水稻幼苗生长及部分抗氧化代谢的影响外文翻译资料
- 用黄体孢子菌沉淀碳酸盐固定有毒金属的体外研究及其在含硫化物尾矿中的应用外文翻译资料
- 水牛妊娠早期生物标志物的编码序列克隆及部分序列密码优化后在大肠杆菌中的表达外文翻译资料


