VIGS—功能基因组学
安妮特贝克尔和马蒂亚斯-兰格
德国不莱梅大学的进化发育遗传学集团、德国不莱梅28359
基因组和EST测序工程对于农学和系统发育中重要的植物物种产生了丰富的序列信息资源,其中大部分物种是很难进行功能基因组学研究的。病毒诱导的基因沉默(VIGS)为不适合稳定遗传转化的物种提供了一个强大的工具。最新的方法允许利用VIGS作为一种高通量的方法,将进一步利用基因组和转录组工程的潜力。
非模式植物物种的功能基因组学
大规模的测序工作为研究基因组结构的进化或植物谱系中的重复事件提供了基础数据。这些新的数据也极大地辅助基因的发现和了解基因功能的发展铺平了道路,植物与病原菌互作,生物合成和发育途径。然而,遗传工具在很大程度上制约了模式植物拟南芥(Arabidopsis thaliana arabidop SIS等),水稻(Oryza sativum),或烟草(Nicotiana tabacum),从而对在植物王国中的基因功能研究的分析方法十分有限。特别是重现性好的稳定的遗传转化是对建立植物生长非常具有挑战性的。因此有可能沉默特定基因及功能研究的快速流失是一个功能强大的技术,它已成功地应用在一个广泛的物种了。在这里,我们强调最近的进展,利用VIGS作为首选工具,在越来越多的植物基因功能中分析的传统方法,如单床作物玉米非常费力的功能基因组学(玉米),大麦(大麦属),小麦(小麦)和双子叶作物植物(葡萄),甜椒(辣椒)、大豆(甘氨酸最大值)。
沉默的分子机制
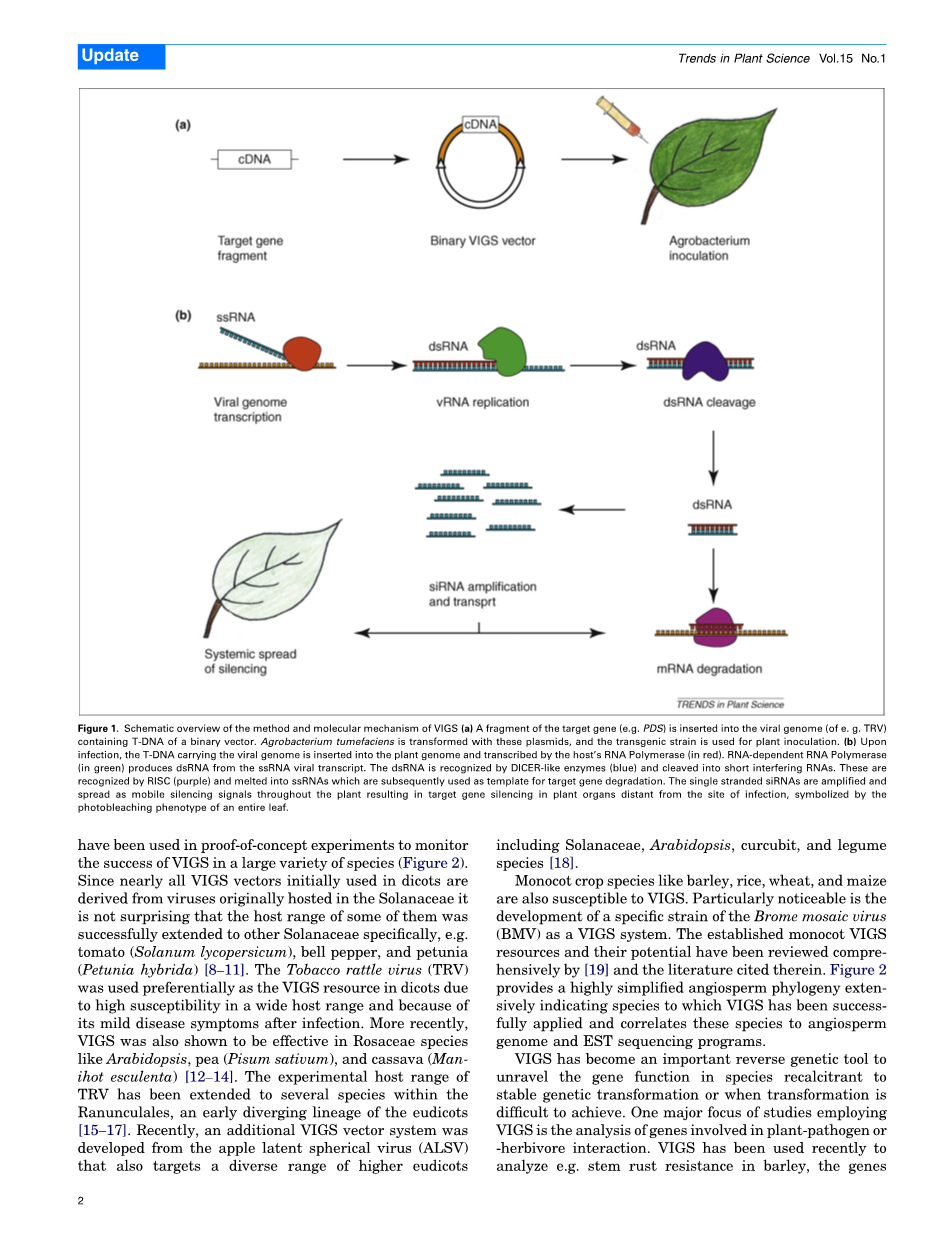
这项技术利用了VIGS重组病毒特异性来降低内源基因的活性,是基于转录后基因沉默(PTGS)[ 1 ]。常用病毒载体基因操纵宿主植物的靶基因片段。植物利用PTGS的先天抗病毒防线抵抗病毒的增殖,采用RNA降解机制,与RNA干扰的途径非常相似。载体的VIGS是标准的二元Ti质粒衍生的载体。用于农杆菌介导的植物变换中插有一个病毒基因组的一部分,病毒基因组内插入多克隆位点添加到宿主植物的内源靶基因(图1A)。200到1300 bp的靶向基因和缺乏聚区中部的片段,如VIGS中polyA尾巴足以在烟草中生长本泌安娜[ 2 ]。植物接种病毒载体通过根癌土壤杆菌感染实现的,含有病毒基因组的T-DNA整合到含有至少一个细胞的宿主基因组中转录和翻译。这导致了双链RNA(dsRNA)的生产从病毒单链RNA模板组装成病毒单链RNA二级结构或互补序列来自于较长的阳性和阴性病毒单链RNA链[ 3]。这些病毒的dsRNA Dicer-蛋白切割成短的干扰RNA(siRNA)双链的约21–24核苷酸(nt)的长度。这些siRNA又合并为单链RNA分子,为RISC(RNA诱导的沉默复合物)检查和破坏与siRNA 互补的RNA[4,5]。在特殊情况下,植物病毒RNA的靶基因mRNA裂解。病毒衍生的沉默信号进一步放大和扩散到整个植物中。siRNA约21个核苷酸的长度被认为是介导短距离运输和RNA依赖的RNA 聚合酶6(RDR6)是远程运输所需的,可能通过放大的沉默信号(对于最近的审查的沉默信号的传输见[ 6 ])。这种系统性的沉默信号在植物的病毒粒子运动中传播发生。当沉默应用于易感植物,寄主植物的靶基因mRNA就在工厂大部退化(图1B)。
VIGS技术成功应用
最初,应对转基因RNA病毒载体基因沉默在烟草中的观察。一个参与类胡萝卜素生物合成途径的沉默基因—八氢番茄红素脱氢酶(PDS),将部分cDNA结合成混合病毒载体组成烟草花叶病毒和番茄花叶病毒沉默的序列。反义RNA病毒传递PDS导致类胡萝卜素合成的抑制作用,这种抑制作用扩散在整个植物中。光保护类胡萝卜素水平降低导致氧化从而导致白叶表型可以很形象地表现出来[ 7]。下面就从1995 PDS基因同源物已用于概念验证实验,在种类繁多的物种中成功监测VIGS的最初实验(图2)。因为最初几乎所有的用于双子叶植物的VIGS是来自于茄科病毒,它们中的一些寄主范围延生到了其他茄科植物中,例如番茄(茄科),甜椒,和矮牵牛(矮牵牛)[ 8,11 ]。因为烟草花叶病毒(TRV)在双子叶植物中广泛的高敏感性和温和的疾病感染症状。最近,沉默也被证明是有效的蔷薇科植物,如拟南芥、豌豆(豌豆),木薯(木薯)[12–14]。实验寄主范围的TRV已扩展到一些早期不同谱系的双子叶植物物种中[ 15–17 ]。最近,另一个VIGS载体系统是从苹果中开发潜在的球形病毒(ALSV),也针对多种高等植物包括茄科、拟南芥,葫芦脲和豆科植物[ 18 ]。
图1。该方法和VIGS(a)分子机制示意图(一)的靶基因片段(如PDS)插入到病毒基因组中(例如TRV),包含一个双元载体T-DNA。农杆菌介导法是用这些质粒转化,而转基因菌株用于植物接种。(b)感染后,T-DNA携带病毒基因组插入到宿主植物基因组,然后转录成RNA聚合酶(红色)。依赖RNA的RNA聚合酶(绿色)产生dsRNA的ssRNA病毒转录体。dsRNA被Dicer样酶识别(蓝色)然后裂解成短干扰RNA。这些都是公认的RISC(紫色)和融入ssRNAs随后被用来作为靶基因降解的模板。单链siRNA沉默信号放大和传播的移动导致靶基因沉默在植物器官的远离部位感染,象征着整个叶片的光漂白表型。
单子叶作物如大麦、大米、小麦、玉米也容易沉默。特别引人注目的是雀麦花叶病毒菌株(BMV)作为一个特定的VIGS系统的发展。建立植物资源及其潜在的单子叶植物进行了全面的文献引证[ 19 ]。图2提供了一个简化的被子植物系统中发育广泛的指示物种,VIGS已成功应用于这些物种的被子植物相关的基因组和EST测序计划中。沉默已成为一种重要的反向遗传工具,能揭示物种基因功能转换,所以物种不能稳定遗传转化是很难实现。此研究的重点是利用VIGS技术分析有关的基因在植物病原体或草食动物中的变化。而最近已被用于分析大麦,如抗秆锈病,参与疾病基因或甲基雅月信号在烟草食动物攻击时的变化[ 20–22]。这也是在进化发育遗传学领域的一项重要的新工具。最近的工作包括研究豌豆根瘤共生基因的一种重要的表征,花发育的B类基因的异常科退化,雄蕊的花器官的贡献,谱系特异性基因功能的收购和像心皮发育调节因子在被子植物进化过程中的演化[ 15,17,23 ]。
然而,在大多数情况下,它所有的持续时间是特定物种的出现时间的沉默效果是短暂的,如大麦条纹花叶病毒(毒)诱导VIGS影响大麦一到两个星期,TRV介导的加利福尼亚罂粟沉默(Eschscholzia夜蛾),在16周后消失,而苹果潜在的球形病毒(ALSV)介导的效应影响能保持大豆植物的整个生命周期,甚至传给下一代[24-26]。另一个挑战就是这项技术的不同表现型在营养生长和生殖组织上需要大量的植物进行筛选表型表型。沉默效应常在所有的植物或植物器官中被发现[24]。
图2。被子植物系统发育基因组测序项目和技术相结合的应用。简化树代表被子植物亲缘关系。从网站获得了被子植物系统发育树的拓扑结构(http://www.mobot.org/mobot/research/apweb)已在紊乱的家庭秩序,水平,用箭头表示属物种的所有。点突出主要植物谱系中绘制的树,绿色方框代表物种与编译的超过20个序列的数据。橙盒标记的物种的基因组正在测序,蓝色框表示与全基因组测序的物种。(http://www.ncbi.nlm.nih.gov/genomes/plants/plantlist。HTML)。成功的目标通过VIGS物种标记为粗体,那些星号实验是容易TRV感染的[ 27 ]。一个完整的物种成功地通过VIGS引用列表网上提供的补充材料。
结束语和观点
VIGS今已应用于被子植物系统学中的30多个植物物种。在大多数情况下,额外的基因功能的直观指标如PDS已经确定。图2高光物种的基因组或转录组测序是广泛的,但对于植物亲缘关系稳定的遗传转化上绝大多数是不定期实现的。一些植物组织如茄科、几种毛茛、葫芦目的证明是容易的途径(一个完整的参考文献列表,参见补充材料)。然而,对于其他的植物如被子植物或其他茄科植物,大的DNA序列集合已经可用,但到目前为止基因功能分析的重现性方法仍无法建立。基因沉默可能是一种有价值的技术资源,在分析基因功能的生物不需要建立稳定的遗传转化协议,有几种物种基因组的数据[27],特别是金鱼(snapdragon)),向日葵(sunflower)、蓖麻(castor-oil plant),红三叶草(red clover),扁豆(haricot or garden bean)、花生(peanut)、萝卜(radish),或洋葱(onion)就属于这一类,因此基因沉默是一种很好的候选物种基因功能的分析技术。可能进一步扩大物种容易使VIGS范围变广,其作为新的载体系统可以从已知的植物病毒中产生。
如果一个稳定的目标基因敲下来会产生表型,那么VIGS是一种快速的方法,可快速测试物种是稳定的遗传转化的,但却是非常耗费时间的。VIGS也可以应用于简便的研究,如研究胚胎致死基因的功能是作用在发育较晚的植物中,在大多数情况下还没有发送到微小或大孢子植物中[ 24 ]。
这项技术未来可能在高通量基因功能分析系统中对于高感品种更易沉默,除了松科植物和加利福尼亚罂粟。结扎自由载体系统已经可以获得[ 10 ],VIGS载体将目的基因的部分序列沉默是是足够有效的[ 2 ]。正向遗传学的屏幕可以在大的非模式植物中插入突变、基因组图谱,但还没有实现,EST序列分析项目提供了足够信息的转录组。为选择最有发展前景的地区的高质量基因组或转录组数据的成绩单提供了一个良好的基础,以避免或诱导同时沉默密切相关的基因序列。出版的高通量方法的例子包括归一化或者消减在VIGS载体中的cDNA文库,随后的植物接种的目的是减少尼古丁水平的克隆、分析所需的外接动力基因介导抗性烟草,并筛选有发育缺陷的番茄[2,29]。这些高通量平台会在扩大我们在进化发育生物学研究方面对于生态研究的新兴领域,他们将运用于评估靶基因的非生物胁迫的抗性,有用的病原菌之间的相互作用然后提高作物育种产量。
致谢
我们感谢罗伯特埃德曼手稿上有帮助的意见和显着提高语言和风格的稿件编辑。A.这些实验室的工作是由德国研究基金资助。因为空间限制,我们特别向那些没有被引用的人表示歉意。
附录A补充资料
与本文相关的辅助数据可以发现,在在线版,在10.1016/j.tplants.2009 DOI。9.002。
参考文献
- Baulcombe, D.C. (1999) Fast forward genetics based on virus-induced gene silencing. Curr. Opin. Plant. Biol. 2, 109–113
- Liu, E. and Page, J.E. (2008) Optimized cDNA libraries for virus-induced gene silencing (VIGS) using tobacco rattle virus. Plant Methods 4, 5
- Donaire, L. et al. (2008) Structural and genetic requirements for the biogenesis of tobacco rattle virus-derived small interfering RNAs. J. Virol. 82, 5167–5177
- Waterhouse, P.M. and Fusaro, A.F. (2006) Plant science. Viruses face a double defense by plant small RNAs. Science 313, 54–55
- Ding, S.W. and Voinnet, O. (2007) Antiviral immunity directed by small RNAs. Cell 130, 413–426
- Kalantidis, K. et al. (2008) RNA silencing movement in plants. Biol. Cell 100, 13–26
- Kumagai, M.H. et al. (1995) Cytoplasmic inhibition of carotenoid biosynthesis with virus-derived RNA. Proc. Natl. Acad. Sci. U. S. A. 92, 1679–1683
- Chung, E. et al. (2004) A Method of High Frequency Virus-induced Gene Silencing in Chili Pepper (Capsicum annuum L. cv. Bukang).
Mol. Cells 17, 377–380
- Brigneti, G. et al. (2004) Virus-induced gene silencing in Solanum species. Plant J. 39, 264–272
-
Liu, Y. et al. (2002) Virus-induced gene silencing in
剩余内容已隐藏,支付完成后下载完整资料

英语原文共 4 页,剩余内容已隐藏,支付完成后下载完整资料
资料编号:[286447],资料为PDF文档或Word文档,PDF文档可免费转换为Word
您可能感兴趣的文章
- 纳米材料介导镉污染土壤原位修复的机理及研究进展外文翻译资料
- 纳米零价铁磁洗受镉污染水稻土壤的有效性验证外文翻译资料
- 镉对水稻叶片生长和养分运输的短期影响外文翻译资料
- 间接竞争ELISA的研制及可视化基于金纳米棒生长的多色酶联免疫吸附试验玉米赤霉烯酮的测定外文翻译资料
- 铅(Pb)诱导的小麦根系生化和超微结构变化外文翻译资料
- 用蚕豆根尖微核试验评估污水灌溉区和生物修复区土壤的遗传毒性外文翻译资料
- 镧在红土壤中的生物累积和其对玉米幼苗生长的影响外文翻译资料
- 铈对水稻幼苗生长及部分抗氧化代谢的影响外文翻译资料
- 用黄体孢子菌沉淀碳酸盐固定有毒金属的体外研究及其在含硫化物尾矿中的应用外文翻译资料
- 水牛妊娠早期生物标志物的编码序列克隆及部分序列密码优化后在大肠杆菌中的表达外文翻译资料


